DNA ambiental - Environmental DNA

| Parte de uma série sobre |
| Código de barras de DNA |
|---|

|
| Por táxons |
|
| De outros |
O DNA ambiental ou eDNA é o DNA coletado de uma variedade de amostras ambientais, como solo , água do mar , neve ou até mesmo do ar, em vez de uma amostra direta de um organismo individual. À medida que vários organismos interagem com o meio ambiente, o DNA é expelido e se acumula em seus arredores de várias fontes. Fontes de exemplo de eDNA incluem, mas não estão limitadas a, fezes , muco , gametas , pele descartada , carcaças e cabelo . Essas amostras podem ser analisadas por métodos de sequenciamento de DNA de alto rendimento , conhecidos como metagenômica , metabarcoding e detecção de uma única espécie, para monitoramento e medição rápidos da biodiversidade . Para melhor diferenciar os organismos dentro de uma amostra, o metabarcoding do DNA é usado no qual a amostra é analisada e usa bibliotecas de DNA previamente estudadas, como o BLAST , para determinar quais organismos estão presentes.
Metabarcoding de eDNA é um novo método de avaliação da biodiversidade em que as amostras são retiradas do ambiente via água, sedimento ou ar do qual o DNA é extraído e, em seguida, amplificado usando primers gerais ou universais na reação em cadeia da polimerase e sequenciado usando sequenciamento de próxima geração para gerar milhares a milhões de leituras. A partir desses dados, a presença de espécies pode ser determinada e a biodiversidade geral avaliada. É um método interdisciplinar que reúne ecologia tradicional baseada em campo com métodos moleculares aprofundados e ferramentas computacionais avançadas.
A análise de eDNA tem grande potencial, não apenas para monitorar espécies comuns, mas para detectar geneticamente e identificar outras espécies existentes que podem influenciar os esforços de conservação. Este método permite o biomonitoramento sem exigir a coleta do organismo vivo, criando a capacidade de estudar organismos invasivos, evasivos ou ameaçados de extinção, sem introduzir estresse antropogênico no organismo. O acesso a essas informações genéticas é uma contribuição crítica para a compreensão do tamanho da população, distribuição das espécies e dinâmica populacional para espécies não bem documentadas. É importante ressaltar que o eDNA costuma ser mais econômico em comparação aos métodos tradicionais de amostragem. A integridade das amostras de eDNA depende de sua preservação no meio ambiente.
Solo, permafrost , água doce e água do mar são macroambientes bem estudados dos quais amostras de eDNA foram extraídas, cada um dos quais incluindo muitos mais subambientes condicionados . Devido à sua versatilidade, o eDNA é aplicado em muitos subambientes , como amostragem de água doce, amostragem de água do mar, amostragem de solo terrestre (tundra permafrost), amostragem de solo aquático (rio, lago, lagoa e sedimento oceânico) ou outros ambientes onde os procedimentos normais de amostragem podem tornar-se problemático.
Visão geral
DNA ambiental ou eDNA descreve o material genético presente em amostras ambientais, como sedimentos, água e ar, incluindo células inteiras, DNA extracelular e organismos potencialmente inteiros. O eDNA pode ser capturado de amostras ambientais e preservado, extraído, amplificado, sequenciado e categorizado com base em sua sequência. A partir dessas informações, é possível a detecção e classificação das espécies. O eDNA pode vir da pele, muco, saliva, esperma, secreções, ovos, fezes, urina, sangue, raízes, folhas, frutas, pólen e corpos em decomposição de organismos maiores, enquanto os microorganismos podem ser obtidos em sua totalidade. A produção de eDNA depende da biomassa, idade e atividade alimentar do organismo, bem como da fisiologia, história de vida e uso do espaço.
Apesar de ser um método relativamente novo de levantamento, o eDNA já provou ter um enorme potencial no monitoramento biológico. Os métodos convencionais para levantamento de riqueza e abundância são limitados pela identificação taxonômica, podem causar perturbação ou destruição do habitat e podem contar com métodos nos quais é difícil detectar espécies pequenas ou elusivas, tornando assim as estimativas para comunidades inteiras impossíveis. O eDNA pode complementar esses métodos visando espécies diferentes, amostrando uma diversidade maior e aumentando a resolução taxonômica . Além disso, o eDNA é capaz de detectar espécies raras, mas não de determinar informações sobre a qualidade da população, como proporções sexuais e condições corporais, por isso é ideal para complementar os estudos tradicionais. Independentemente disso, ele tem aplicações úteis na detecção das primeiras ocorrências de espécies invasoras, a presença contínua de espécies nativas consideradas extintas ou ameaçadas e outras espécies evasivas ocorrendo em baixas densidades que seriam difíceis de detectar por meios tradicionais.
A degradação do eDNA no meio ambiente limita o escopo dos estudos de eDNA, já que frequentemente apenas pequenos segmentos de material genético permanecem, particularmente em regiões tropicais quentes. Além disso, os vários períodos de degradação com base nas condições ambientais e no potencial do DNA para viajar através da mídia, como a água, podem afetar a inferência de tendências espaço-temporais em escala fina de espécies e comunidades. Apesar dessas desvantagens, o eDNA ainda tem o potencial de determinar a abundância relativa ou de classificação, pois alguns estudos descobriram que ela corresponde à biomassa, embora a variação inerente às amostras ambientais torne sua quantificação difícil. Embora o eDNA tenha inúmeras aplicações em conservação, monitoramento e avaliação de ecossistemas, bem como outras ainda a serem descritas, as concentrações altamente variáveis de eDNA e a heterogeneidade potencial através do corpo d'água tornam essencial que o procedimento seja otimizado, de preferência com um estudo piloto para cada nova aplicação, para garantir que o projeto de amostragem seja apropriado para detectar o alvo.
DNA da comunidade
Embora a definição de eDNA pareça direta, as linhas entre as diferentes formas de DNA tornam-se confusas, particularmente em comparação com o DNA da comunidade , que é descrito como amostras de organismos em massa. Surge uma questão a respeito de microrganismos inteiros capturados em amostras de eDNA: esses organismos alteram a classificação da amostra para uma amostra de DNA da comunidade? Além disso, a classificação do material genético das fezes é problemática e frequentemente chamada de eDNA. A diferenciação entre os dois é importante, pois o DNA da comunidade indica a presença de um organismo em um determinado momento e lugar, enquanto o eDNA pode ter vindo de um local diferente, de fezes de predadores ou de presença anterior; no entanto, essa diferenciação costuma ser impossível. No entanto, o eDNA pode ser vagamente classificado como incluindo muitos setores de pesquisa de biodiversidade de DNA, incluindo análise fecal e amostras em massa quando eles são aplicáveis à pesquisa de biodiversidade e análise de ecossistema.
selfDNA
O conceito de selfDNA decorre de descobertas feitas por cientistas da Universidade de Nápoles Federico II , que foram relatadas durante 2015 na revista New Phytologist ( https://www.newphytologist.org/news/view/97 ), sobre o self- efeito inibitório do DNA extracelular em plantas (), mas também em bactérias, fungos, algas, plantas, protozoários e insetos (). A fonte ambiental de tal DNA extracelular é proposta para ser lixo vegetal, mas também outras fontes em diferentes ecossistemas e organismos, com o tamanho dos fragmentos de DNA experimentalmente demonstrado ter um efeito inibitório sobre seus organismos conspecíficos tipicamente variando entre 200 e 500 pares de bases. O fenômeno selfDNA foi postulado para conduzir interações ecológicas e ser mecanicamente mediado por padrões moleculares associados a danos (DAMPs) (,), e ter potencial para o desenvolvimento de aplicações biocidas (). Veja também Circulating free DNA .
Metabarcoding de eDNA
Em 2019, os métodos de pesquisa de eDNA foram expandidos para poder avaliar comunidades inteiras a partir de uma única amostra. Este processo envolve metabarcoding , que pode ser precisamente definido como o uso de primers de reação em cadeia da polimerase (PCR) geral ou universal em amostras de DNA misturadas de qualquer origem, seguido por sequenciamento de próxima geração (NGS) de alto rendimento para determinar a composição de espécies do amostra. Esse método é comum na microbiologia há anos, mas está apenas encontrando seu fundamento na avaliação de macroorganismos. As aplicações de metabarcodificação de eDNA em todo o ecossistema têm o potencial não apenas de descrever comunidades e biodiversidade, mas também de detectar interações e ecologia funcional em grandes escalas espaciais, embora possa ser limitado por leituras falsas devido a contaminação ou outros erros. Ao todo, o metabarcoding do eDNA aumenta a velocidade, a precisão e a identificação em relação ao código de barras tradicional e diminui o custo, mas precisa ser padronizado e unificado, integrando taxonomia e métodos moleculares para um estudo ecológico completo.
Metabarcoding eDNA tem aplicações para monitoramento de diversidade em todos os habitats e grupos taxonômicos, reconstrução de ecossistemas antigos, interações planta-polinizador , análise de dieta, detecção de espécies invasivas, respostas à poluição e monitoramento da qualidade do ar. Metabarcodificação de eDNA é um método exclusivo ainda em desenvolvimento e provavelmente permanecerá em fluxo por algum tempo, conforme os avanços da tecnologia e os procedimentos se tornem padronizados. No entanto, como o metabarcoding é otimizado e seu uso se torna mais difundido, é provável que se torne uma ferramenta essencial para o monitoramento ecológico e o estudo de conservação global.
DNA extracelular e relíquia
O DNA extracelular, às vezes chamado de DNA relíquia, é o DNA de micróbios mortos. O DNA extracelular nu (eDNA), a maior parte dele liberado pela morte celular, é quase onipresente no meio ambiente. Sua concentração no solo pode ser tão alta quanto 2 μg / L, e sua concentração em ambientes aquáticos naturais pode ser tão alta quanto 88 μg / L. Várias funções possíveis foram propostas para o eDNA: ele pode estar envolvido na transferência horizontal de genes ; pode fornecer nutrientes; e pode atuar como um tampão para recrutar ou titular íons ou antibióticos. O DNA extracelular atua como um componente da matriz extracelular funcional nos biofilmes de várias espécies bacterianas. Pode atuar como um fator de reconhecimento para regular a fixação e a dispersão de tipos específicos de células no biofilme; pode contribuir para a formação de biofilme; e pode contribuir para a força física do biofilme e resistência ao estresse biológico.
Sob o nome de DNA ambiental, o eDNA tem visto um uso crescente nas ciências naturais como uma ferramenta de pesquisa para a ecologia , monitorando os movimentos e a presença de espécies na água, no ar ou na terra e avaliando a biodiversidade de uma área.
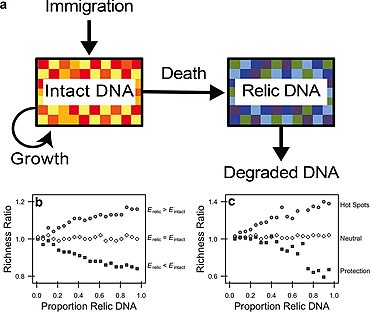
No diagrama à direita, a quantidade de DNA de relíquia em um ambiente microbiano é determinada por entradas associadas à mortalidade de indivíduos viáveis com DNA intacto e por perdas associadas à degradação do DNA de relíquia. Se a diversidade de sequências contidas no pool de DNA da relíquia for suficientemente diferente daquela no pool de DNA intacto, o DNA da relíquia pode distorcer as estimativas da biodiversidade microbiana (conforme indicado por caixas coloridas diferentes) ao amostrar o DNA total (intacto + relíquia) piscina. Dados Padronizados sobre Iniciativas (STARDIT) foram propostos como uma forma de padronizar dados sobre métodos de amostragem e análise, e relações taxonômicas e ontológicas.
RNA ambiental
Coleção
Sedimentos terrestres
A importância da análise de eDNA decorreu do reconhecimento das limitações apresentadas pelos estudos baseados em cultura . Os organismos se adaptaram para prosperar nas condições específicas de seus ambientes naturais. Embora os cientistas trabalhem para imitar esses ambientes, muitos organismos microbianos não podem ser removidos e cultivados em um laboratório. A versão mais antiga desta análise começou com o RNA ribossômico ( rRNA ) em micróbios para entender melhor os micróbios que vivem em ambientes hostis. A composição genética de alguns micróbios só é acessível por meio da análise de eDNA. Técnicas analíticas de eDNA foram inicialmente aplicadas a sedimentos terrestres, produzindo DNA de mamíferos, pássaros, insetos e plantas extintos e existentes. As amostras extraídas desses sedimentos terrestres são comumente referenciadas como 'DNA antigo sedimentar' ( DNA de seda ou DNA de sujeira ). A análise de eDNA também pode ser usada para estudar as comunidades florestais atuais, incluindo tudo, desde pássaros e mamíferos a fungos e vermes. As amostras podem ser obtidas a partir do solo, fezes, 'DNA de mordida' de onde as folhas foram mordidas, plantas e folhas onde os animais foram, e das refeições de sangue de mosquitos capturados que podem ter comido sangue de qualquer animal na área. Alguns métodos também podem tentar capturar células com armadilhas de cabelo e lixa em áreas comumente atravessadas por espécies-alvo.
Sedimentos aquáticos
O sedaDNA foi subsequentemente usado para estudar a diversidade animal antiga e verificado usando registros fósseis conhecidos em sedimentos aquáticos. Os sedimentos aquáticos são privados de oxigênio e, portanto, protegem o DNA da degradação. Além de estudos antigos, essa abordagem pode ser usada para entender a diversidade animal atual com sensibilidade relativamente alta. Enquanto amostras de água típicas podem ter o DNA degradado de forma relativamente rápida, as amostras de sedimentos aquáticos podem ter DNA útil dois meses após a espécie estar presente. Um problema com os sedimentos aquáticos é que não se sabe onde o organismo depositou o eDNA, pois ele poderia ter se movido na coluna d'água.
Aquático (coluna de água)
O estudo do eDNA na coluna d'água pode indicar a composição da comunidade de um corpo d'água. Antes do eDNA, as principais formas de estudar a diversidade de águas abertas eram a pesca e a captura, o que requer recursos como financiamento e mão de obra qualificada, enquanto o eDNA só precisa de amostras de água. Este método é eficaz porque o pH da água não afeta o DNA tanto quanto se pensava, e a sensibilidade pode ser aumentada com relativa facilidade. A sensibilidade é a probabilidade de o marcador de DNA estar presente na água da amostra e pode ser aumentada simplesmente tomando mais amostras, tendo amostras maiores e aumentando o PCR . O eDNA se degrada com relativa rapidez na coluna d'água, o que é muito benéfico em estudos de conservação de curto prazo, como a identificação de espécies presentes.
Pesquisadores da Experimental Lakes Area em Ontário, Canadá e da McGill University descobriram que a distribuição do eDNA reflete a estratificação do lago . À medida que as estações e a temperatura da água mudam, a densidade da água também muda, formando camadas distintas em pequenos lagos boreais no verão e no inverno. Essas camadas se misturam durante a primavera e o outono. O uso do habitat dos peixes está correlacionado à estratificação (por exemplo, peixes de água fria como a truta do lago permanecerão na água fria) e também a distribuição do eDNA, como descobriram esses pesquisadores.

No diagrama acima à esquerda, a linha tracejada rosa indica o uso de um rastreador químico para rastreamento de contaminação durante a testemunhagem. A linha tracejada branca representa o núcleo do sedimento. Pequenos círculos amarelos indicam intervalos de amostragem de sedaDNA teóricos, correspondendo a gráficos de pizza à direita. Os gráficos de torta representam paleo-comunidades hipotéticas detectáveis a partir da análise shotgun de sedaDNA, em que a maioria (~ 75%) das sequências de sedaDNA recuperadas se originam de bactérias e onde sedaDNA de taxa fossilizante / formadora de cisto aumenta em relação a não fossilização / não cisto -formadores de taxa com profundidade submarina (assumindo que sedaDNA de taxa fossilizantes / formadores de cistos se preserva melhor do que os taxa não fossilizantes / não formadores de cistos). O tamanho decrescente dos gráficos de pizza com a profundidade submarina indica uma diminuição esperada no sedaDNA.
Monitorando espécies
O eDNA pode ser usado para monitorar espécies ao longo do ano e pode ser muito útil no monitoramento da conservação. A análise de eDNA foi bem-sucedida na identificação de muitos táxons diferentes de plantas aquáticas, mamíferos aquáticos, peixes, mexilhões, fungos e até mesmo parasitas. O eDNA tem sido usado para estudar espécies enquanto minimiza qualquer interação humana induzindo estresse, permitindo aos pesquisadores monitorar a presença de espécies em escalas espaciais maiores de forma mais eficiente. O uso mais prevalente na pesquisa atual é o uso de eDNA para estudar a localização de espécies em risco, espécies invasoras e espécies-chave em todos os ambientes. O eDNA é especialmente útil para estudar espécies com populações pequenas porque o eDNA é sensível o suficiente para confirmar a presença de uma espécie com relativamente pouco esforço para coletar dados, o que muitas vezes pode ser feito com uma amostra de solo ou água. O eDNA depende da eficiência do sequenciamento e da análise genômica, bem como dos métodos de pesquisa usados, que continuam a se tornar mais eficientes e baratos. Alguns estudos mostraram que o eDNA amostrado de riachos e ambientes costeiros decaiu a um nível indetectável em cerca de 48 horas.
O DNA ambiental pode ser aplicado como uma ferramenta para detectar organismos de baixa abundância nas formas ativa e passiva. Levantamentos de eDNA ativos têm como alvo espécies individuais ou grupos de táxons para detecção usando PCR em tempo real quantitativo e específico para espécies altamente sensíveis ou marcadores digitais de PCR de gotículas . A metodologia CRISPR-Cas também foi aplicada à detecção de espécies únicas de eDNA; utilizando a enzima Cas12a e permitindo maior especificidade ao detectar táxons simpátricos. Pesquisas de eDNA passivas empregam sequenciamento de DNA massivamente paralelo para amplificar todas as moléculas de eDNA em uma amostra sem um alvo a priori em mente, fornecendo evidências de DNA gerais da composição da comunidade biótica.
Declínio de artrópodes terrestres

Os artrópodes terrestres estão experimentando um declínio maciço na Europa e também no mundo, embora apenas uma fração das espécies tenha sido avaliada e a maioria dos insetos ainda não seja descrita pela ciência. Como um exemplo, os ecossistemas de pastagens são o lar de diversos grupos taxonômicos e funcionais de artrópodes terrestres , como polinizadores , insetos fitófagos e predadores, que usam néctar e pólen como fontes de alimento e tecido do caule e folha para alimentação e desenvolvimento. Essas comunidades abrigam espécies ameaçadas de extinção , uma vez que muitos habitats desapareceram ou estão sob ameaça significativa. Portanto, grandes esforços estão sendo realizados para restaurar os ecossistemas de pastagem europeus e conservar a biodiversidade . Por exemplo, polinizadores como abelhas e borboletas representam um importante grupo ecológico que sofreu um declínio severo na Europa, indicando uma perda dramática da biodiversidade das pastagens. A grande maioria das plantas com flores são polinizadas por insetos e outros animais, tanto em regiões temperadas quanto nos trópicos. A maioria das espécies de insetos são herbívoros que se alimentam de diferentes partes das plantas, e a maioria deles são especialistas, contando com uma ou algumas espécies de plantas como principal recurso alimentar. No entanto, dada a lacuna no conhecimento sobre as espécies de insetos existentes, e o fato de que a maioria das espécies ainda não foi descrita, é claro que para a maioria das espécies de plantas no mundo, há conhecimento limitado sobre as comunidades de artrópodes que elas abrigam e com as quais interagem.
As comunidades de artrópodes terrestres têm sido tradicionalmente coletadas e estudadas usando métodos, como armadilhas de Malaise e armadilhas de queda , que são muito eficazes, mas métodos um tanto pesados e potencialmente invasivos. Em alguns casos, essas técnicas ficam aquém da realização de levantamentos eficientes e padronizados, devido, por exemplo, à plasticidade fenotípica , espécies intimamente relacionadas e dificuldades em identificar estágios juvenis. Além disso, a identificação morfológica depende diretamente da perícia taxonômica , que está em declínio. Todas essas limitações do monitoramento tradicional da biodiversidade criaram uma demanda por abordagens alternativas. Enquanto isso, o avanço nas tecnologias de sequenciamento de DNA fornece continuamente novos meios de obtenção de dados biológicos. Portanto, várias novas abordagens moleculares foram recentemente sugeridas para a obtenção de dados rápidos e eficientes sobre comunidades de artrópodes e suas interações por meio de técnicas genéticas não invasivas. Isso inclui a extração de DNA de fontes como amostras em massa ou sopas de insetos, minas de folhas vazias, teias de aranha, fluido de planta de jarro, amostras ambientais como solo e água (DNA ambiental [eDNA]), planta hospedeira e identificação de dieta predatória a partir de extratos de DNA de insetos, e fezes de predadores de morcegos. Recentemente, também o DNA do pólen ligado a insetos foi usado para recuperar informações sobre as interações planta-polinizador . Muitos desses estudos recentes baseiam-se na codificação do DNA - sequenciamento de alto rendimento de amplicons de PCR usando primers genéricos.
Mamíferos
Trilhas de neve
Os pesquisadores da vida selvagem em áreas nevadas também usam amostras de neve para coletar e extrair informações genéticas sobre espécies de interesse. O DNA de amostras de rastros de neve foi usado para confirmar a presença de espécies raras e evasivas como ursos polares, raposas árticas, linces, carcajus e pescadores.
DNA do ar
Em 2021, os pesquisadores demonstraram que o eDNA pode ser coletado do ar e usado para identificar mamíferos.
Gerenciando pescarias
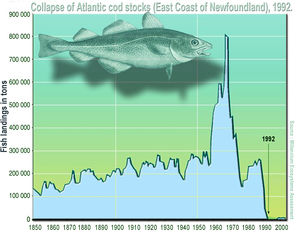
O gerenciamento bem-sucedido da pesca comercial depende de pesquisas padronizadas para estimar a quantidade e distribuição dos estoques pesqueiros . O bacalhau do Atlântico (Gadus morhua) é um exemplo icônico que demonstra como dados mal limitados e tomadas de decisão desinformadas podem resultar em declínio catastrófico dos estoques e consequentes problemas econômicos e sociais. As avaliações tradicionais de estoque de espécies de peixes demersais têm se baseado principalmente em pesquisas com redes de arrasto , que forneceram um fluxo valioso de informações para os tomadores de decisão. No entanto, existem algumas desvantagens notáveis nos levantamentos com redes de arrasto demersal, incluindo custo, seletividade / capturabilidade das artes, destruição de habitat e cobertura restrita (por exemplo, ambientes de fundo de substrato duro, áreas marinhas protegidas).
O DNA ambiental (eDNA) surgiu como uma alternativa potencialmente poderosa para estudar a dinâmica do ecossistema. A constante perda e derramamento de material genético de macroorganismos confere uma pegada molecular em amostras ambientais que podem ser analisadas para determinar a presença de espécies-alvo específicas ou caracterizar a biodiversidade. A combinação de sequenciamento de última geração e amostragem de eDNA foi aplicada com sucesso em sistemas aquáticos para documentar padrões espaciais e temporais na diversidade da fauna de peixes. Para desenvolver ainda mais a utilidade do eDNA para o gerenciamento das pescas, compreender a capacidade das quantidades de eDNA de refletir a biomassa dos peixes no oceano é um próximo passo importante.
Relações positivas entre quantidades de eDNA e biomassa e abundância de peixes foram demonstradas em sistemas experimentais. No entanto, as variações conhecidas entre a produção de eDNA e as taxas de degradação podem complicar essas relações em sistemas naturais. Além disso, em sistemas oceânicos, grandes volumes de habitat e fortes correntes podem resultar na dispersão física de fragmentos de DNA para longe dos organismos-alvo. Esses fatores de confusão foram considerados anteriormente para restringir a aplicação de monitoramento quantitativo de eDNA em ambientes oceânicos.
Apesar dessas restrições potenciais, vários estudos em ambientes marinhos encontraram relações positivas entre as quantidades de eDNA e esforços de pesquisa complementares, incluindo marcação de rádio, pesquisas visuais, eco-sondagem e pesquisas com redes de arrasto. No entanto, os estudos que quantificam as concentrações de eDNA alvo de espécies de peixes comerciais com pesquisas padronizadas de arrasto em ambientes marinhos são muito mais escassos. Neste contexto, comparações diretas de concentrações de eDNA com biomassa e métricas de avaliação de estoque, como captura por unidade de esforço (CPUE), são necessárias para entender a aplicabilidade do monitoramento de eDNA para contribuir com os esforços de gestão da pesca.
Sedimentos do fundo do mar
O DNA extracelular na superfície dos sedimentos do fundo do mar é de longe o maior reservatório de DNA dos oceanos do mundo. As principais fontes de DNA extracelular em tais ecossistemas são representadas pela liberação in situ de DNA de organismos bentônicos mortos e / ou outros processos, incluindo lise celular devido à infecção viral, exsudação celular e excreção de células viáveis, decomposição de vírus e entradas alóctones do coluna de água. Estudos anteriores forneceram evidências de que uma fração importante do DNA extracelular pode escapar dos processos de degradação, permanecendo preservada nos sedimentos. Esse DNA representa, potencialmente, um repositório genético que registra processos biológicos que ocorrem ao longo do tempo.
Investigações recentes revelaram que o DNA preservado em sedimentos marinhos é caracterizado por um grande número de sequências de genes altamente diversas. Em particular, o DNA extracelular tem sido usado para reconstruir a diversidade procariótica e eucariótica passada em ecossistemas bentônicos caracterizados por baixas temperaturas e / ou condições permanentemente anóxicas.
O diagrama à direita mostra a rede OTU ( unidade taxonômica operacional ) dos reservatórios de DNA extracelular dos sedimentos das diferentes margens continentais. O tamanho do ponto dentro da rede é proporcional à abundância de sequências para cada OTU. Os pontos circulados em vermelho representam OTUs de núcleo extracelular, os pontos circulados em amarelo são parcialmente compartilhados (entre dois ou mais pools) OTUs, os pontos circulados em preto são OTUs exclusivos de cada pool. As OTUs principais contribuindo com pelo menos 20 sequências são mostradas. Os números entre parênteses representam o número de conexões entre OTUs e amostras: 1 para OTUs exclusivos, 2–3 para OTUs parcialmente compartilhados e 4 para OTUs principais.
Estudos anteriores sugeriram que a preservação do DNA também pode ser favorecida em sistemas bentônicos caracterizados por altas entradas de matéria orgânica e taxas de sedimentação, como margens continentais. Esses sistemas, que representam ca. 15% do fundo do mar global também são pontos críticos de diversidade procariótica bentônica e, portanto, podem representar locais ideais para investigar a diversidade procariótica preservada no DNA extracelular.
A distribuição espacial da diversidade procariótica tem sido intensamente estudada em ecossistemas bentônicos de alto mar por meio da análise de "DNA ambiental" (isto é, o material genético obtido diretamente de amostras ambientais sem quaisquer sinais óbvios de material de origem biológica). No entanto, não se sabe até que ponto as sequências de genes contidas no DNA extracelular podem alterar as estimativas da diversidade das assembleias procarióticas atuais.
DNA antigo sedimentar
|
−4500 -
-
-
-
−4000 -
-
-
-
-3500 -
-
-
-
-3000 -
-
-
-
-2500 -
-
-
-
-2000 -
-
-
-
-1500 -
-
-
-
-1000 -
-
-
-
−500 -
-
-
-
0 -
|
|
|
||||||||||||||||||||||||||||||||||||||||||
As análises de DNA antigo preservado em vários arquivos transformaram a compreensão da evolução das espécies e dos ecossistemas. Embora estudos anteriores tenham se concentrado no DNA extraído de amostras taxonomicamente restritas (como ossos ou tecido congelado), os avanços em sequenciamento de alto rendimento e bioinformática agora permitem a análise de DNA antigo extraído de arquivos sedimentares, o chamado sedaDNA. A acumulação e preservação do sedaDNA enterrado em sedimentos terrestres e lacustres tem sido objeto de pesquisa e interpretação ativa. No entanto, estudar a deposição de DNA no fundo do oceano e sua preservação em sedimentos marinhos é mais complexo porque o DNA tem que viajar por uma coluna de água por vários quilômetros. Ao contrário do ambiente terrestre, com transporte difuso de biomassa subfóssil da terra, a maior parte do sedaDNA marinho é derivada da comunidade planctônica , que é dominada por micróbios marinhos e protistas marinhos . Após a morte do plâncton superficial, seu DNA está sujeito a um transporte através da coluna d'água, durante a qual grande parte da matéria orgânica associada é consumida e respirada . Este transporte pode demorar entre 3 a 12 dias dependendo do tamanho e morfologia do teste. No entanto, ainda não está claro como exatamente o eDNA planctônico, definido como o DNA total presente no ambiente depois, sobrevive a esse transporte, se a degradação ou transporte estão associados à classificação ou advecção lateral e, finalmente, se o eDNA que chega ao fundo do mar é preservado em sedimentos marinhos sem maiores distorções de sua composição.
Apesar da longa exposição à degradação sob condições óxidas durante o transporte na coluna de água e concentração substancialmente mais baixa de matéria orgânica no fundo do mar, há evidências de que o eDNA planctônico é preservado em sedimentos marinhos e contém sinais ecológicos exploráveis. Estudos anteriores mostraram a preservação do sedaDNA em sedimentos marinhos depositados sob anóxia com quantidades anormalmente altas de matéria orgânica preservada, mas investigações posteriores indicam que o sedaDNA também pode ser extraído de sedimentos marinhos normais, dominados por frações minerais clásticas ou biogênicas . Além disso, a baixa temperatura da água do mar profundo (0–4 ° C) garante uma boa preservação do sedaDNA. Usando foraminíferos planctônicos como uma "Pedra de Roseta", permitindo a comparação de assinaturas de sedaDNA por testes fósseis co-ocorrentes desses organismos, Morard et al. mostraram em 2017 que a impressão digital do plâncton eDNA que chega ao fundo do mar preserva a assinatura ecológica desses organismos em grande escala geográfica. Isso indica que o eDNA da comunidade planctônica é depositado no fundo do mar abaixo, junto com agregados, esqueletos e outro material planctônico que afunda. Se isso for verdade, o sedaDNA deve ser capaz de registrar assinaturas da hidrografia da superfície do oceano, afetando a composição das comunidades do plâncton, com a mesma resolução espacial dos restos de esqueleto do plâncton. Além disso, se o eDNA do plâncton chega ao fundo do mar em associação com agregados ou conchas, é possível que resista ao transporte através da coluna de água por fixação em superfícies minerais. O mesmo mecanismo foi proposto para explicar a preservação do sedaDNA em sedimentos, sugerindo que o fluxo de eDNA planctônico encapsulado em teste de calcita chegando ao fundo do mar está condicionado à preservação no momento do soterramento.
O sedaDNA do foraminífero planctônico é um substituto ideal tanto "horizontalmente" para avaliar a resolução espacial da reconstrução das características hidrográficas oceânicas da superfície passada, quanto "verticalmente", para rastrear inequivocamente o soterramento de seu sinal ao longo da coluna de sedimentos. Na verdade, o fluxo de eDNA foraminíferos planctônicos deve ser proporcional ao fluxo de conchas foraminíferas mortas que afundam no fundo do mar, permitindo um benchmarking independente do sinal de eDNA. O eDNA é uma ferramenta poderosa para estudar ecossistemas porque não requer conhecimento taxonômico direto, permitindo, assim, que informações sejam coletadas sobre todos os organismos presentes em uma amostra, mesmo no nível críptico . No entanto, a atribuição das sequências de eDNA a organismos conhecidos é feita por meio da comparação com sequências de referência (ou códigos de barras ) disponibilizados em repositórios públicos ou bancos de dados com curadoria. A taxonomia dos foraminíferos planctônicos é bem compreendida [1] </ref> e existem códigos de barras que permitem o mapeamento quase completo de amplicons de eDNA na taxonomia baseada na morfologia do teste foraminífero. É importante ressaltar que a composição das comunidades de foraminíferos planctônicos está intimamente ligada à hidrografia de superfície e este sinal é preservado por testes de fósseis depositados no fundo do mar. Uma vez que o eDNA foraminífero acumulado no sedimento do oceano pode ser recuperado, ele pode ser usado para analisar as mudanças nas comunidades planctônicas e bentônicas ao longo do tempo.
Pesquisa participativa e ciência cidadã
A relativa simplicidade da amostragem de eDNA se presta a projetos que buscam envolver as comunidades locais como parte de projetos de pesquisa, incluindo a coleta e análise de amostras de DNA. Isso pode capacitar as comunidades locais (incluindo os povos indígenas) a se envolverem ativamente no monitoramento das espécies em um ambiente e ajudar a tomar decisões informadas como parte do modelo de pesquisa de ação participativa. Um exemplo de tal projeto foi demonstrado pela instituição de caridade Ciência para Todos com o projeto 'Wild DNA'.
Veja também
Referências
Outras referências
- Schallenberg, Lena; Wood, Susie A .; Pochon, Xavier; Pearman, John K. (2020). "O que o DNA no meio ambiente pode nos dizer sobre um ecossistema?" . Fronteiras para jovens mentes . 7 . doi : 10.3389 / frym.2019.00150 . S2CID 210714520 .