RAD52 - RAD52
Homólogo RAD52 (S. cerevisiae) , também conhecido como RAD52 , é uma proteína que em humanos é codificada pelo gene RAD52 .
Função
A proteína codificada por este gene compartilha similaridade com a Saccharomyces cerevisiae Rad52, uma proteína importante para o reparo de quebra de fita dupla de DNA e recombinação homóloga . Este produto do gene foi mostrado para ligar extremidades de DNA de fita simples e mediar a interação DNA-DNA necessária para o anelamento de fitas de DNA complementares . Também foi descoberto que ele interage com a proteína de recombinação de DNA RAD51 , o que sugere seu papel na recombinação e reparo de DNA relacionado a RAD51.
Papel no reparo de recombinação de DNA
RAD52 medeia a função de RAD51 em reparo recombinacional homólogo (HRR) em ambas as leveduras Saccharomyces cerevisiae e em células de mamíferos de camundongos e humanos. No entanto, a proteína RAD52 tem funções distintas no HRR de leveduras e humanos. Em S. cerevisae , a proteína Rad52, agindo sozinha, facilita o carregamento da proteína Rad51 em DNA de fita simples pré-revestido com proteína A de replicação na fase pré - sináptica de recombinação.
Em camundongos e humanos, no entanto, BRCA2 medeia principalmente a montagem ordenada de RAD51 em ssDNA, a forma que é ativa para emparelhamento homólogo e invasão de fita. BRCA2 também redireciona RAD51 de dsDNA e impede a dissociação de ssDNA. Além disso, os quatro parálogos de RAD51 , consistindo em RAD51B ( RAD51L1 ), RAD51C (RAD51L2), RAD51D ( RAD51L3 ), XRCC2 , formam um complexo denominado complexo BCDX2. Este complexo participa do recrutamento ou estabilização do RAD51 nos locais de dano. O complexo BCDX2 parece agir facilitando a montagem ou estabilidade do filamento da nucleoproteína RAD51 . No entanto, na presença de uma mutação BRCA2, o RAD52 humano pode mediar a montagem do RAD51 no ssDNA e substituir o BRCA2 no reparo do DNA de recombinação homóloga , embora com menor eficiência do que o BRCA2.
Além disso, o RAD52 humano, em combinação com ERCC1 , promove a via de reparo de DNA homólogo propenso a erros de recozimento de fita simples. Embora propensa a erros, esta via de reparo pode ser necessária para a sobrevivência de células com danos no DNA que não são reparáveis de outra forma.
O RAD52 humano também tem um papel importante no reparo de quebras de fita dupla de DNA em locais de transcrição ativos durante a fase G0 / G1 do ciclo celular. O reparo dessas quebras de fita dupla parece usar um mecanismo de recombinação baseado em modelo de RNA dependente de RAD52. A proteína B da síndrome de Cockayne (CSB) (codificada por ERCC6 ) localiza-se em quebras de fita dupla em locais de transcrição ativa, seguida por RAD51 , RAD51C e RAD52 para realizar o reparo de recombinação homóloga usando o RNA recém-sintetizado como modelo.
microRNAs e risco de câncer
Três regiões primárias não traduzidas (3'UTRs) de RNAs mensageiros (mRNAs) geralmente contêm sequências regulatórias que podem causar silenciamento de RNA pós-transcricional . Essas 3'-UTRs geralmente contêm locais de ligação para microRNAs (miRNAs). Ao se ligar a locais específicos dentro da 3'-UTR, os miRNAs podem diminuir a expressão gênica de vários mRNAs, tanto inibindo a tradução quanto causando diretamente a degradação do transcrito.
MicroRNAs (miRNAs) parecem regular a expressão de mais de 60% dos genes codificadores de proteínas do genoma humano. Um microRNA, miR-210 , reprime RAD52. Conforme observado por Devlin et al., MiR-210 é regulado positivamente na maioria dos tumores sólidos e afeta negativamente o resultado clínico.
A 3'-UTR de RAD52 também possui um sítio de ligação para o microRNA let-7 . Mulheres com um polimorfismo de nucleotídeo único (SNP) no local de ligação para let-7 (rs7963551), que causa ligação reduzida de let-7, provavelmente têm expressão aumentada de RAD52 (como foi mostrado para este SNP no fígado). Mulheres com este SNP no 3'UTR de RAD52 mostraram um risco reduzido de câncer de mama com um odds ratio de 0,84, intervalo de confiança de 95% de 0,75-0,95.
Em uma população chinesa Han, o mesmo SNP acima no local de ligação 3'-UTR do RAD52 para let-7 (rs7963551) reduziu o risco de glioma. O risco de glioma associado ao genótipo RAD52 rs7963551 teve uma razão de chances (em comparação com aqueles sem SNP) de 0,44 para aqueles com mais de 41 anos e uma razão de chances de 0,58 para aqueles com 41 anos ou mais jovens.
Li et al. encontraram uma diminuição significativa do risco de carcinoma celular hepático entre indivíduos com o genótipo RAD52 rs7963551 CC (o mesmo SNP acima) em comparação com aqueles com o genótipo AA em uma população chinesa. Eles também descobriram que em 44 amostras de tecido hepático humano normal, a presença do SNP rs7963551 foi associada a um aumento significativo da expressão de mRNA de RAD52.
Assim, a expressão aumentada de RAD52 é protetora contra vários tipos de câncer.
Outro estudo de sítios de ligação de microRNA alterados em RAD52 e seus efeitos na suscetibilidade ao câncer foi realizado por Naccarati et al. Eles descobriram dois locais de ligação de microRNA RAD52 que foram frequentemente alterados e tiveram um efeito sobre o risco de câncer de cólon. Indivíduos com um SNP homozigoto ou heterozigoto em rs1051669 estavam em risco aumentado de câncer de cólon (OR 1,78, IC 95% 1,13-2,80, p = 0,01 para homozigotos e OR 1,72, IC 95% 1,10-2,692, p = 0,02 para heterozigotos). Portadores heterozigotos do outro SNP RAD52 (rs11571475) estavam em risco reduzido de câncer de cólon (OR 0,76, IC de 95% 0,58-1,00, p = 0,05). De 21 genes na via de reparo de recombinação homóloga e 7 genes na via de junção de extremidade não homóloga examinada, os únicos SNPs encontrados em regiões de ligação de microRNA que estavam em frequência alta o suficiente para avaliar e que afetaram os riscos de câncer de cólon, foram os dois em RAD52 e um em MRE11A .
O dano ao DNA parece ser a principal causa subjacente do câncer, e deficiências no reparo do DNA parecem ser a base de muitas formas de câncer. Se o reparo do DNA for deficiente, o dano ao DNA tende a se acumular. Esse excesso de dano ao DNA pode aumentar os erros mutacionais durante a replicação do DNA devido à síntese de translesão propensa a erros . O excesso de dano ao DNA também pode aumentar as alterações epigenéticas devido a erros durante o reparo do DNA. Essas mutações e alterações epigenéticas podem dar origem ao câncer . O aumento frequente induzido por microRNA ou deficiência de reparo de DNA mediado por RAD52 devido a alterações de ligação de microRNA provavelmente contribui para a prevenção ou progressão de câncer de mama, cérebro, fígado ou cólon.
Interações
RAD52 demonstrou interagir com RAD51. O Rad52 irá facilitar o carregamento de Rad51 no ssDNA interferindo com a proteína RPA .
Complementação intragênica
Quando múltiplas cópias de um polipeptídeo codificado por um gene formam um agregado, esta estrutura de proteína é referida como um multímero. Quando um multímero é formado a partir de polipeptídeos produzidos por dois alelos mutantes diferentes de um determinado gene, o multímero misto pode exibir maior atividade funcional do que os multímeros não misturados formados por cada um dos mutantes isoladamente. Nesse caso, o fenômeno é denominado complementação intragênica . A Saccharomyces cerevisiae RAD52 alelo mutante que expressa uma proteína truncada C-terminal foi encontrada para complementar outras RAD52 mutantes missense alelos. Este achado de complementação intragênica sugere que a proteína RAD52 tem uma estrutura multimérica que permite interações cooperativas entre os monômeros constituintes.
Referências
Leitura adicional
- Muris DF, Bezzubova O, Buerstedde JM, Vreeken K, Balajee AS, Osgood CJ, Troelstra C, Hoeijmakers JH, Ostermann K, Schmidt H (novembro de 1994). "Clonagem de genes humanos e de camundongo homólogos a RAD52, um gene de levedura envolvido no reparo e recombinação do DNA" . Pesquisa de mutação . 315 (3): 295–305. doi : 10.1016 / 0921-8777 (94) 90040-x . hdl : 1765/3072 . PMID 7526206 .
- Shen Z, Denison K, Lobb R, Gatewood JM, Chen DJ (janeiro de 1995). "Os homólogos humanos e de camundongo do gene RAD52 de levedura: clonagem de cDNA, análise de sequência, atribuição ao cromossomo humano 12p12.2-p13 e expressão de mRNA em tecidos de camundongo" . Genômica . 25 (1): 199–206. doi : 10.1016 / 0888-7543 (95) 80126-7 . PMID 7774919 .
- Park MS (junho de 1995). "A expressão de RAD52 humano confere resistência à radiação ionizante em células de mamíferos" . The Journal of Biological Chemistry . 270 (26): 15467–70. doi : 10.1074 / jbc.270.26.15467 . PMID 7797537 .
- Shen Z, Pardington-Purtymun PE, Comeaux JC, Moyzis RK, Chen DJ (Set 1996). "UBL1, uma proteína semelhante à ubiquitina humana associada às proteínas RAD51 / RAD52 humanas" . Genômica . 36 (2): 271–9. doi : 10.1006 / geno.1996.0462 . PMID 8812453 .
- Shen Z, Pardington-Purtymun PE, Comeaux JC, Moyzis RK, Chen DJ (Out 1996). "Associações de UBE2I com proteínas RAD52, UBL1, p53 e RAD51 em um sistema de dois híbridos de levedura" . Genômica . 37 (2): 183–6. doi : 10.1006 / geno.1996.0540 . PMID 8921390 .
- Chen G, Yuan SS, Liu W, Xu Y, Trujillo K, Song B, Cong F, Goff SP, Wu Y, Arlinghaus R, Baltimore D, Gasser PJ, Park MS, Sung P, Lee EY (abril de 1999). "Montagem induzida por radiação do complexo de recombinação Rad51 e Rad52 requer ATM e c-Abl" (PDF) . The Journal of Biological Chemistry . 274 (18): 12748–52. doi : 10.1074 / jbc.274.18.12748 . PMID 10212258 . S2CID 2587580 .
- Kito K, Wada H, Yeh ET, Kamitani T (dezembro de 1999). "Identificação de novas isoformas de RAD52 humano". Biochimica et Biophysica Acta (BBA) - Estrutura e expressão gênica . 1489 (2–3): 303–14. doi : 10.1016 / s0167-4781 (99) 00214-6 . PMID 10673031 .
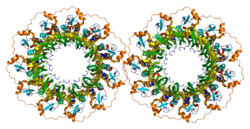
- Stasiak AZ, Larquet E, Stasiak A, Müller S, Engel A, Van Dyck E, West SC, Egelman EH (março de 2000). "A proteína Rad52 humana existe como um anel heptamérico" . Biologia atual . 10 (6): 337–40. doi : 10.1016 / S0960-9822 (00) 00385-7 . PMID 10744977 . S2CID 16087838 .
- Parsons CA, Baumann P, Van Dyck E, West SC (agosto de 2000). "Ligação precisa de terminais de DNA de fita simples pela proteína RAD52 humana" . The EMBO Journal . 19 (15): 4175–81. doi : 10.1093 / emboj / 19.15.4175 . PMC 306603 . PMID 10921897 .
- Mer G, Bochkarev A, Gupta R, Bochkareva E, Frappier L, Inglês CJ, Edwards AM, Chazin WJ (outubro de 2000). "Base estrutural para o reconhecimento de proteínas de reparo de DNA UNG2, XPA e RAD52 pelo fator de replicação RPA" . Cell . 103 (3): 449–56. doi : 10.1016 / S0092-8674 (00) 00136-7 . PMID 11081631 . S2CID 16640087 .
- Ranatunga W, Jackson D, Flowers II RA, Borgstahl GE (julho de 2001). "A proteína RAD52 humana tem extrema estabilidade térmica". Bioquímica . 40 (29): 8557–62. doi : 10.1021 / bi0155089 . PMID 11456495 .
- Van Dyck E, Stasiak AZ, Stasiak A, West SC (outubro de 2001). "Visualização de intermediários de recombinação produzidos por anelamento de fita única mediado por RAD52" . Relatórios EMBO . 2 (10): 905–9. doi : 10.1093 / embo-reports / kve201 . PMC 1084079 . PMID 11571269 .
- Kim PM, Allen C, Wagener BM, Shen Z, Nickoloff JA (novembro de 2001). "Superexpressão de RAD51 e RAD52 humanos reduz a recombinação homóloga induzida por quebra de fita dupla em células de mamíferos" . Nucleic Acids Research . 29 (21): 4352–60. doi : 10.1093 / nar / 29.21.4352 . PMC 60192 . PMID 11691922 .
- Yáñez RJ, Porter AC (fevereiro de 2002). "Os efeitos diferenciais da superexpressão de Rad52p no direcionamento de genes e recombinação homóloga extracromossômica em uma linha de células humanas" . Nucleic Acids Research . 30 (3): 740–8. doi : 10.1093 / nar / 30.3.740 . PMC 100286 . PMID 11809887 .
- Jackson D, Dhar K., Wahl JK, Wold MS, Borgstahl GE (agosto de 2002). "Análise da proteína de replicação humana A: complexo Rad52: evidência de diafonia entre RPA32, RPA70, Rad52 e DNA". Journal of Molecular Biology . 321 (1): 133–48. doi : 10.1016 / S0022-2836 (02) 00541-7 . PMID 12139939 .
- Kagawa W, Kurumizaka H, Ishitani R, Fukai S, Nureki O, Shibata T, Yokoyama S (agosto de 2002). "Estrutura de cristal do domínio de emparelhamento homólogo da recombinase Rad52 humana na forma undecamérica" . Molecular Cell . 10 (2): 359–71. doi : 10.1016 / S1097-2765 (02) 00587-7 . PMID 12191481 .
- Singleton MR, Wentzell LM, Liu Y, West SC, Wigley DB (outubro de 2002). "Estrutura do domínio de recozimento de fita única da proteína RAD52 humana" . Anais da Academia Nacional de Ciências dos Estados Unidos da América . 99 (21): 13492–7. doi : 10.1073 / pnas.212449899 . PMC 129701 . PMID 12370410 .
- Liu J, Meng X, Shen Z (outubro de 2002). "Associação da proteína RAD52 humana com fatores de transcrição". Comunicações de pesquisa bioquímica e biofísica . 297 (5): 1191–6. doi : 10.1016 / S0006-291X (02) 02353-7 . PMID 12372413 .
- Han J, Hankinson SE, De Vivo I, Colditz GA, Hunter DJ (outubro de 2002). "Nenhuma associação entre um polimorfismo de códon de parada em RAD52 e o risco de câncer de mama". Epidemiologia do câncer, biomarcadores e prevenção . 11 (10 Pt 1): 1138–9. PMID 12376524 .
- Kitao H, Yuan ZM (dezembro de 2002). "Regulação da formação de focos nucleares de Rad52 induzida por radiação ionizante por fosforilação mediada por c-Abl" . The Journal of Biological Chemistry . 277 (50): 48944–8. doi : 10.1074 / jbc.M208151200 . PMID 12379650 .