Aconitase - Aconitase
| aconitato hidratase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
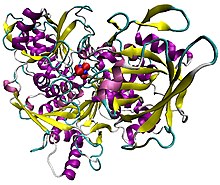 Ilustração de aconitase suína em complexo com o aglomerado [Fe 4 S 4 ]. A proteína é colorida pela estrutura secundária, e os átomos de ferro são azuis e o enxofre vermelho.
| |||||||||
| Identificadores | |||||||||
| EC nº | 4.2.1.3 | ||||||||
| CAS no. | 9024-25-3 | ||||||||
| Bancos de dados | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrada BRENDA | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | Entrada KEGG | ||||||||
| MetaCyc | via metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBsum | ||||||||
| Ontologia Genética | AmiGO / QuickGO | ||||||||
| |||||||||
| Família Aconitase (aconitate hidratase) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura da aconitase.
| |||||||||
| Identificadores | |||||||||
| Símbolo | Aconitase | ||||||||
| Pfam | PF00330 | ||||||||
| InterPro | IPR001030 | ||||||||
| PRÓSITO | PDOC00423 | ||||||||
| SCOP2 | 1aco / SCOPe / SUPFAM | ||||||||
| |||||||||
Aconitase (aconitato hidratase; EC 4.2.1.3 ) é uma enzima que catalisa a estereo-especifica de isomerização de citrato a isocitrato através de cis - aconitato no ciclo do ácido tricarboxílico , um não- redox processo -active.
Estrutura
Aconitase, apresentada nas estruturas da margem direita desta página, apresenta duas estruturas ligeiramente diferentes, consoante se encontre activada ou desactivada. Na forma inativa, sua estrutura é dividida em quatro domínios. Contando a partir do terminal N , apenas os três primeiros desses domínios estão envolvidos em interações próximas com o cluster [3Fe-4S], mas o local ativo consiste em resíduos de todos os quatro domínios, incluindo o domínio C-terminal maior . O cluster Fe-S e um ânion SO 4 2 também residem no site ativo. Quando a enzima é ativada, ela ganha um átomo de ferro adicional, criando um cluster [4Fe-4S]. No entanto, a estrutura do resto da enzima permanece quase inalterada; os átomos conservados entre as duas formas estão essencialmente nas mesmas posições, até uma diferença de 0,1 angstroms.
Função
Em contraste com a maioria das proteínas ferro-enxofre que funcionam como transportadores de elétrons, o grupo ferro-enxofre da aconitase reage diretamente com um substrato enzimático. Aconitase tem um cluster ativo [Fe 4 S 4 ] 2+ , que pode se converter em uma forma inativa [Fe 3 S 4 ] + . Foi demonstrado que três resíduos de cisteína (Cys) são ligantes do centro [Fe 4 S 4 ]. No estado ativo, o íon de ferro lábil do aglomerado [Fe 4 S 4 ] não é coordenado por Cys, mas por moléculas de água.
A proteína de ligação ao elemento responsivo ao ferro (IRE-BP) e a 3-isopropilmalato desidratase (α-isopropilmalato isomerase; EC 4.2.1.33 ), uma enzima que catalisa a segunda etapa na biossíntese da leucina , são homólogas da aconitase conhecidas. Os elementos reguladores de ferro (IREs) constituem uma família de estruturas de haste-alça não codificantes de 28 nucleotídeos que regulam o armazenamento de ferro, a síntese de heme e a absorção de ferro. Eles também participam da ligação ao ribossomo e controlam o turnover (degradação) do mRNA . A proteína reguladora específica, a IRE-BP, liga-se a IREs nas regiões 5 'e 3', mas apenas ao RNA na forma apo, sem o cluster Fe-S. A expressão de IRE-BP em células cultivadas revelou que a proteína funciona como uma aconitase ativa, quando as células estão repletas de ferro, ou como uma proteína ligadora de RNA ativa, quando as células estão depletadas de ferro. IRE-BPs mutantes, nos quais qualquer um ou todos os três resíduos Cys envolvidos na formação de Fe-S são substituídos por serina , não têm atividade de aconitase, mas retêm propriedades de ligação a RNA.
A conitase é inibida pelo fluoroacetato , portanto, o fluoroacetato é venenoso. O fluoroacetato, no ciclo do ácido cítrico, pode entrar inocentemente como fluorocitrato. No entanto, a aconitase não consegue se ligar a este substrato e, portanto, o ciclo do ácido cítrico é interrompido. O aglomerado de ferro e enxofre é altamente sensível à oxidação por superóxido .
Mecanismo
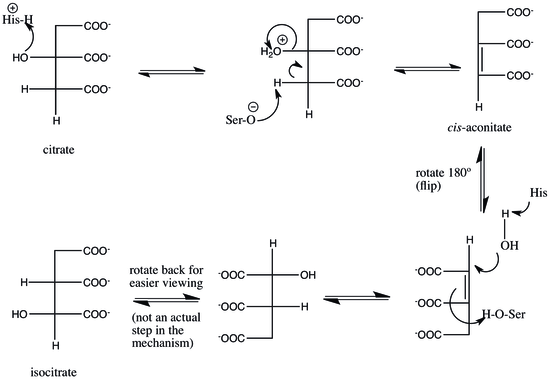
Aconitase emprega um mecanismo de desidratação-hidratação. Os resíduos catalíticos envolvidos são His-101 e Ser-642. His-101 protona o grupo hidroxila em C3 de citrato, permitindo que ele saia como água, e Ser-642 simultaneamente abstrai o próton em C2, criando uma ligação dupla entre C2 e C3, e formando o denominado intermediário cis- aconitado ( os dois grupos carboxila na ligação dupla são cis ). O átomo de carbono do qual o hidrogênio é removido é aquele que veio do oxaloacetato na etapa anterior do ciclo do ácido cítrico, não aquele que veio da acetil CoA , embora esses dois carbonos sejam equivalentes, exceto que um é " pró- R "e o outro" pró- S "(ver Prociralidade ). Neste ponto, o intermediário é girado 180 °. Essa rotação é conhecida como "flip". Por causa dessa inversão, diz-se que o intermediário passa de um "modo citrato" para um "modo isocitrato".
Como exatamente essa inversão ocorre é discutível. Uma teoria é que, na etapa de limitação da taxa do mecanismo, o cis- aconitado é liberado da enzima e, em seguida, reconectado no modo isocitrato para completar a reação. Esta etapa de limitação de taxa garante que a estereoquímica correta , especificamente (2R, 3S), seja formada no produto final. Outra hipótese é que o cis- aconitado permanece ligado à enzima enquanto passa do modo citrato para o isocitrato.
Em qualquer dos casos, a inversão do cis- aconitado permite que as etapas de desidratação e hidratação ocorram nas faces opostas do intermediário. A conitase catalisa a eliminação / adição trans de água, e o flip garante que a estereoquímica correta seja formada no produto. Para completar a reação, os resíduos de serina e histidina revertem suas ações catalíticas originais: a histidina, agora básica, abstrai um próton da água, preparando-o como um nucleófilo para atacar em C2, e a serina protonada é desprotonada pelo duplo cis- aconitar ligação para completar a hidratação, produzindo isocitrato.
Membros da família
Aconitases são expressas em bactérias para humanos. Os humanos expressam as seguintes duas isozimas de aconitase :
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
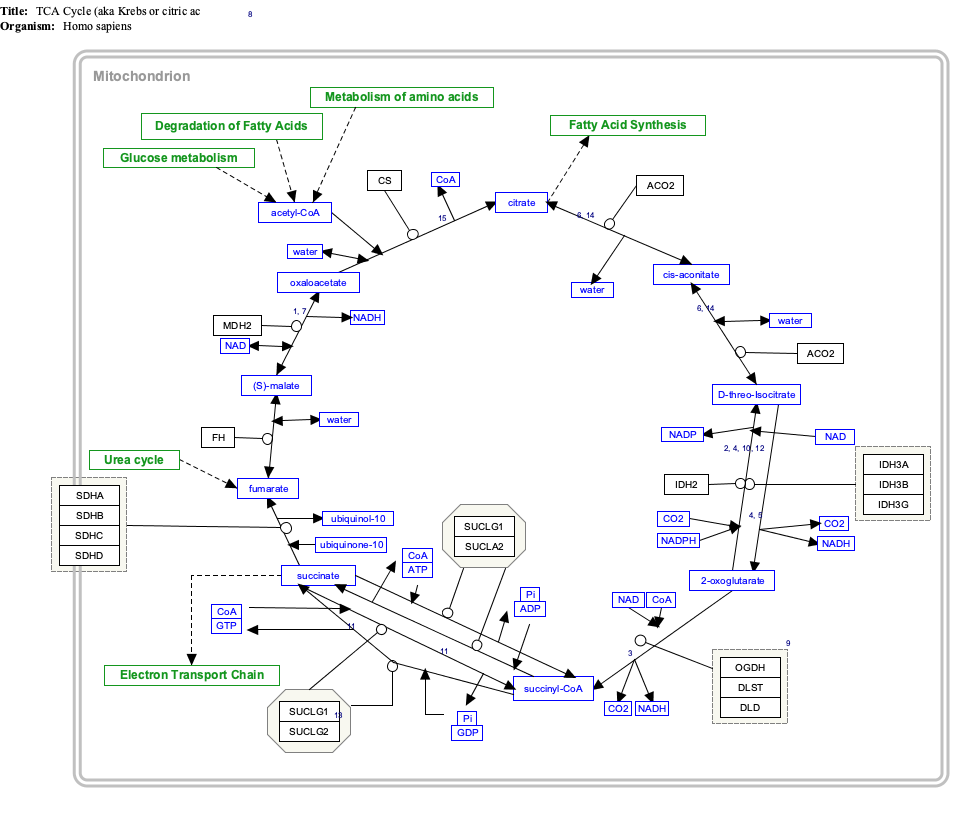
Mapa de caminho interativo
Clique nos genes, proteínas e metabólitos abaixo para acessar os respectivos artigos.
Referências
Leitura adicional
- Frishman D, Hentze MW (julho de 1996). "Conservação de resíduos de aconitase revelada por análise de sequência múltipla. Implicações para relações de estrutura / função" . European Journal of Biochemistry / FEBS . 239 (1): 197–200. doi : 10.1111 / j.1432-1033.1996.0197u.x . PMID 8706708 .
links externos
- Aconitase na Biblioteca Nacional de Medicina dos Estados Unidos, cabeçalhos de assuntos médicos (MeSH)
- Proteopedia Aconitase - a estrutura Aconitase em 3D interativo