DMSO redutase - DMSO reductase
| Dimetilsulfóxido redutase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Número CE | 1.8.5.3 | ||||||||
| Bancos de dados | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrada BRENDA | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | Entrada KEGG | ||||||||
| MetaCyc | via metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
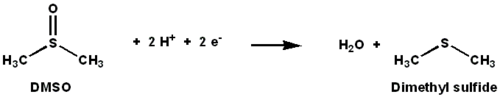
A DMSO redutase é uma enzima contendo molibdênio que catalisa a redução de dimetilsulfóxido (DMSO) a dimetilsulfeto (DMS). Esta enzima atua como redutase terminal em condições anaeróbicas em algumas bactérias, com DMSO sendo o aceptor de elétrons terminal. Durante o curso da reação, o átomo de oxigênio em DMSO é transferido para o molibdênio e, em seguida, reduzido a água.
A DMSO redutase (DMSOR) e outros membros da família da DMSO redutase são exclusivos de bactérias e arquéias . Enzimas desta família na fosforilação oxidativa anaeróbia e respiração litotrófica baseada em doadores inorgânicos . Essas enzimas foram projetadas para degradar oxoanions. O DMSOR catalisa a transferência de dois elétrons e um átomo de oxigênio na reação: O sítio ativo do DMSOR contém molibdênio, que é raro na biologia.
Estrutura terciária e site ativo
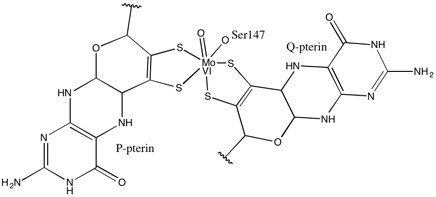
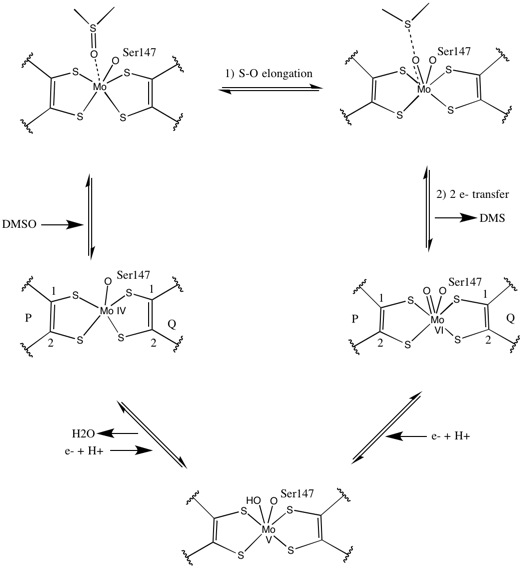
Quanto a outros membros da família da DMSO redutase, a estrutura terciária do DMSOR é composta de domínios I-IV circundantes de Mo, com o domínio IV interagindo fortemente com cofatores de Mo de piranopterinditioleno (P- e Q-pterina) do sítio ativo. Os membros da família da DMSO redutase diferem em termos de seus sítios ativos. No caso do DMSOR, o centro de Mo é encontrado em dois ditiolenos fornecidos por dois cofatores de piranopterina. Esses cofatores orgânicos, chamados molibdopterinas , estão ligados ao GMP para criar uma forma de dinucleotídeo. Um quinto ligante semelhante a cap adicional é a cadeia lateral O do resíduo de serina-147, classificando posteriormente a enzima como DMSO redutase do Tipo III. A serina InType I e II é substituída por resíduos de cisteína e aspartato, respectivamente. Dependendo do estado redox do Mo, que flutua entre IV, V ou VI conforme a reação progride, o núcleo de Mo do sítio ativo também pode ser ligado a um átomo de oxigênio de um grupo aqua-, hidroxo- ou oxo, respectivamente . Estudos demonstraram que a identidade particular do aminoácido usado para coordenar o núcleo de Mo influencia muito o potencial de ponto médio redox de Mo e o estado de protonação da ligação do grupo de oxigênio, que são determinantes-chave no mecanismo da enzima para catálise.
Mecanismo
Estudos iniciais com DMSO 18 isotópico estabeleceram um mecanismo de dupla oxotransferase para DMSOR de R. sphaeroides . Neste mecanismo, o O 18 marcado é transferido do substrato para o Mo, que então transfere o O 18 para 1,3,5-triaza-7-fosfadamantano (PTA) para produzir PTAO 18 . Em um mecanismo análogo, o DMSO transfere O para Mo, e o centro de Mo (VI) O resultante é reduzido, produzindo água.
Estudos sobre complexos de Mo-bis- ditioleno sintéticos sugerem que sejam transferência de oxigênio, transferência de elétrons. Usando S K-edge XAS e DFT, esses estudos de modelo apontam para cisão SO combinada e transferência de elétrons. As taxas são proporcionais à diminuição da força de ligação do substrato XO e ao aumento da afinidade de prótons do substrato.
A cristalografia de raios-X estabeleceu que a estrutura terciária geral da enzima permanece constante durante a progressão da reação. No entanto, vários experimentos diferentes conduzidos em DMSOR de R. sphaeroides relataram resultados diferentes para a atividade de coordenação dos quatro potenciais ligantes de ditioleno. Enquanto uma investigação de cristalografia de raios-X concluiu a coordenação equidistante de todos os quatro ligantes Mo-S na forma oxidada, o que é apoiado por numerosos estudos de espectroscopia de absorção de raios-X (XAS), um estudo diferente caracterizou as distâncias Mo-S assimétricas. Ambos os estudos, bem como estudos de ressonância paramagnética eletrônica (EPR), previram que o sítio ativo de Mo é altamente flexível em termos de posição e grau de potenciais coordenadas do ligante.
Os dados que sugeriram dois cofatores de piranopterina significativamente assimétricos foram usados para propor um mecanismo de reação. Na forma Mo VI totalmente oxidada do sítio ativo, o grupo oxo e os ligantes da serina foram coordenados a 1,7 A de distância do centro de Mo. S1 e S2 da P-pterina e S1 da Q-pterina foram localizados 2,4 A de distância do Mo, e S2 da Q-pterina foi localizado a 3,1 A de distância. Esta assimetria de pterina pode ser o resultado do efeito trans do grupo oxo enfraquecendo a ligação S2-Mo, que está localizada diretamente oposta ao grupo oxo.
Em contraste, a estrutura da forma totalmente reduzida de Mo IV do sítio ativo mostrou que S1 e S2 P-pterina e S1 Q-pterina mantiveram a coordenação total, no entanto, S2 da Q-pterina se afastou do centro do metal, indicando diminuição da coordenação . Esta mudança no comprimento da ligação ligante-Mo é consistente com o mecanismo proposto de transferência direta de oxigênio do substrato DMSO para o Mo. Uma coordenação ditioleno mais fraca na forma de enzima reduzida pode facilitar a ligação direta do S = O. Na redução de Mo e protonação do grupo oxo, é proposto que uma fonte de elétrons de citocromo poderia se ligar a uma depressão acima do sítio ativo e reduzir diretamente o centro de Mo, ou alternativamente, este citocromo poderia se ligar a uma alça polipeptídica bem solvatada na proximidade da Q-pterina, e a Q-pterina poderia mediar esta transferência de elétrons.
Localização e regulação celular
Em R. sphaeroides , DMSOR é uma proteína solúvel em água de subunidade única que não requer cofatores adicionais além da pterina. Em E. coli , o DMSOR está embutido na membrana e tem três subunidades únicas, uma das quais inclui o cofator pterina característico, outra que contém quatro clusters 4Fe: 4S e uma subunidade transmembrana final que se liga e oxida o menaquinol. A transferência de um e- do menaquinol para os clusters 4Fe: 4S e finalmente para o sítio ativo pterin-Mo gera um gradiente de prótons usado para a geração de ATP.
DMSOR regulado predominantemente em um nível transcricional. É codificado pelo gene dor e expresso quando ativado por uma cascata de sinal, que está sob a regulação das proteínas DorS, DorR e DorC. Um estudo de fusões lacZ (genes repórter) para os promotores dorS, dorR e dorC correspondentes concluiu que a expressão de DorR e DorC aumentou em ambientes de oxigênio reduzido, mas a expressão de DorS não foi afetada pela concentração de oxigênio. A expressão de DorC também aumentou com o aumento das concentrações de DMSO.
Impacto ambiental
O DMS, um produto do DMSOR, é um componente do ciclo do enxofre . O DMS é oxidado a metanossulfonatos , que nuclea a condensação de nuvens sobre oceanos abertos, onde a fonte alternativa de nucleação, a poeira, está ausente. A formação de nuvens é um componente chave no aumento do albedo da Terra e na regulação da temperatura atmosférica, portanto, essa enzima e a reação que ela catalisa podem ser úteis na fronteira do controle do clima.