Selenocisteína - Selenocysteine
|
|
| Nomes | |
|---|---|
|
Nome IUPAC
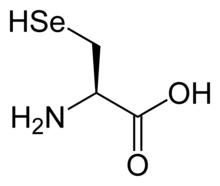
Ácido 2-amino-3-selanilpropanóico
|
|
| Outros nomes
L -Selenocisteína; 3-selanil- L- alanina; Selênio cisteína
|
|
| Identificadores | |
|
Modelo 3D ( JSmol )
|
|
| ChEBI | |
| ChEMBL | |
| ChemSpider | |
| DrugBank | |
| ECHA InfoCard |
100.236.386 |
| KEGG | |
|
PubChem CID
|
|
| UNII | |
|
Painel CompTox ( EPA )
|
|
|
|
|
|
| Propriedades | |
| C 3 H 7 N O 2 Se | |
| Massa molar | 168,065 g · mol −1 |
|
Exceto onde indicado de outra forma, os dados são fornecidos para materiais em seu estado padrão (a 25 ° C [77 ° F], 100 kPa). |
|
|
|
|
| Referências da Infobox | |
Selenocisteína (símbolo Sec ou U , em publicações mais antigas também como Se-Cys ) é o 21º aminoácido proteinogênico . As selenoproteínas contêm resíduos de selenocisteína. A selenocisteína é um análogo da cisteína mais comum com selênio no lugar do enxofre .
A selenocisteína está presente em várias enzimas (por exemplo, glutationa peroxidases , tetraiodotironina 5 ′ desiodinases , tiorredoxina redutases , formato desidrogenases , glicina redutases , selenofosfato sintetase 2 , metionina- R- sulfóxido redutase B1 ( SEPX1 ) e algumas hidrogenases B1 ( SEPX1 ). Ocorre nos três domínios da vida .
A selenocisteína foi descoberta pela bioquímica Thressa Stadtman no National Institutes of Health .
Química
A selenocisteína é o análogo Se da cisteína. Raramente é encontrado (e não está disponível comercialmente) porque é muito suscetível à oxidação do ar. Mais comum é o derivado oxidado de selenocistina, que possui uma ligação Se-Se. Tanto a selenocisteína quanto a selenocistina são sólidos brancos. O grupo Se-H é mais ácido ( p K a = 5,43) do que o grupo tiol, portanto é desprotonado em pH fisiológico .
Estrutura
A selenocisteína tem uma estrutura semelhante à da cisteína , mas com um átomo de selênio no lugar do enxofre usual. Ele tem um grupo selenol . Como outros aminoácidos proteinogênicos naturais, a cisteína e a selenocisteína têm quiralidade L na notação D / L mais antiga com base na homologia com o D - e L - gliceraldeído . No sistema R / S mais recente de designar quiralidade, com base nos números atômicos de átomos próximos ao carbono assimétrico, eles têm quiralidade R , devido à presença de enxofre ou selênio como um segundo vizinho ao carbono assimétrico. Os aminoácidos quirais restantes, tendo apenas átomos mais leves nessa posição, têm quiralidade S. )
As proteínas que contêm um resíduo de selenocisteína são chamadas de selenoproteínas . A maioria das selenoproteínas contém um único resíduo de selenocisteína. As selenoproteínas que exibem atividade catalítica são chamadas de selenoenzimas.
Biologia
A selenocisteína tem um potencial de redução menor do que a cisteína. Essas propriedades o tornam muito adequado em proteínas que estão envolvidas na atividade antioxidante .
Embora seja encontrado nos três domínios da vida, não é universal em todos os organismos. Ao contrário de outros aminoácidos presentes nas proteínas biológicas , a selenocisteína não é codificada diretamente no código genético . Em vez disso, ele é codificado de uma maneira especial por um códon UGA , que normalmente é o códon de parada "opala" . Tal mecanismo é denominado recodificação translacional e sua eficiência depende da selenoproteína que está sendo sintetizada e dos fatores de iniciação da tradução . Quando as células são cultivadas na ausência de selênio, a tradução das selenoproteínas termina no códon UGA, resultando em uma enzima truncada e não funcional. O códon UGA é feito para codificar a selenocisteína pela presença de uma sequência de inserção de selenocisteína (SECIS) no mRNA . O elemento SECIS é definido por sequências de nucleotídeos características e padrões de emparelhamento de bases de estrutura secundária. Em bactérias , o elemento SECIS está normalmente localizado imediatamente após o códon UGA dentro do quadro de leitura para a selenoproteína. Em Archaea e em eucariotos , o elemento SECIS está na região 3 'não traduzida (3' UTR) do mRNA e pode direcionar múltiplos códons UGA para codificar resíduos de selenocisteína.
Ao contrário dos outros aminoácidos, nenhum pool livre de selenocisteína existe na célula. Sua alta reatividade causaria danos às células. Em vez disso, as células armazenam selênio na forma oxidada menos reativa, selenocistina, ou na forma metilada, selenometionina. A síntese de selenocisteína ocorre em um tRNA especializado , que também funciona para incorporá-lo a polipeptídeos nascentes.
A estrutura primária e secundária do tRNA específico da selenocisteína, tRNA Sec , difere daquelas dos tRNAs padrão em vários aspectos, mais notavelmente por ter uma haste aceitadora de 8 pares de bases (bactérias) ou 10 pares de bases (eucariotos), um braço de região variável longa e substituições em várias posições de base bem conservadas. Os tRNAs de selenocisteína são inicialmente carregados com serina pela seril-tRNA ligase , mas o Ser-tRNA Sec resultante não é usado para tradução porque não é reconhecido pelo fator de alongamento de tradução normal ( EF-Tu em bactérias, eEF1A em eucariotos).
Em vez disso, o resíduo de seril ligado ao tRNA é convertido em um resíduo de selenocisteína pela enzima selenocisteína sintase contendo fosfato de piridoxal . Em eucariotos e arquéias, duas enzimas são necessárias para converter o resíduo de seril ligado ao tRNA em resíduo de selenocisteinil do tRNA: PSTK ( O - fosfoseril -tRNA [Ser] Sec quinase) e selenocisteína sintase. Finalmente, o Sec-tRNA Sec resultante é especificamente ligado a um fator de alongamento de tradução alternativo (SelB ou mSelB (ou eEFSec)), que o entrega de forma direcionada aos ribossomos que traduzem mRNAs para selenoproteínas. A especificidade deste mecanismo de entrega é provocada pela presença de um domínio de proteína extra (em bactérias, SelB) ou uma subunidade extra ( SBP2 para mSelB / eEFSec eucariótico) que se ligam às estruturas secundárias de RNA correspondentes formadas pelos elementos SECIS na selenoproteína mRNAs.
A selenocisteína é decomposta pela enzima selenocisteína liase em L - alanina e seleneto.
Desde 2021, 136 proteínas humanas (em 37 famílias) são conhecidas por conter selenocisteína (selenoproteínas).
Os derivados da selenocisteína γ-glutamil- Se- metilselenocisteína e Se- metilselenocisteína ocorrem naturalmente em plantas dos gêneros Allium e Brassica .
Formulários
As aplicações biotecnológicas da selenocisteína incluem o uso de Sec marcado com 73 Se (meia-vida de 73 Se = 7,2 horas) em estudos de tomografia por emissão de pósitrons (PET) e Sec marcado com 75 Se (meia-vida de 75 Se = 118,5 dias) em específico radiomarcação , facilitação da determinação de fase por difração anômala de comprimento de onda múltiplo em cristalografia de raios-X de proteínas pela introdução de Sec sozinho, ou Sec junto com selenometionina (SeMet), e incorporação do isótopo 77 Se estável, que tem um spin nuclear de1/2e pode ser usado para RMN de alta resolução , entre outros.
Veja também
- Pirrolisina , outro aminoácido que não está no conjunto básico de 20.
- Selenometionina , outro aminoácido contendo selênio, que é aleatoriamente substituído por metionina.
Referências
Leitura adicional
- Zinoni F, Birkmann A, Stadtman TC, Böck A (julho de 1986). "Sequência de nucleotídeos e expressão do polipeptídeo contendo selenocisteína de formato desidrogenase (ligado a formato-hidrogênio-liase) de Escherichia coli" . Anais da Academia Nacional de Ciências dos Estados Unidos da América . 83 (13): 4650–4. Bibcode : 1986PNAS ... 83.4650Z . doi : 10.1073 / pnas.83.13.4650 . PMC 323799 . PMID 2941757 .
- Zinoni F, Birkmann A, Leinfelder W, Böck A (maio de 1987). "Inserção cotranslacional de selenocisteína em formato desidrogenase de Escherichia coli dirigida por um códon UGA" . Anais da Academia Nacional de Ciências dos Estados Unidos da América . 84 (10): 3156–60. Bibcode : 1987PNAS ... 84.3156Z . doi : 10.1073 / pnas.84.10.3156 . PMC 304827 . PMID 3033637 .
- Cone JE, Del Río RM, Davis JN, Stadtman TC (agosto de 1976). "Caracterização química do componente selenoproteína da redutase de glicina clostridial: identificação da selenocisteína como a porção organo-selênio" . Anais da Academia Nacional de Ciências dos Estados Unidos da América . 73 (8): 2659–63. Bibcode : 1976PNAS ... 73.2659C . doi : 10.1073 / pnas.73.8.2659 . PMC 430707 . PMID 1066676 .
- Fenyö D, Beavis RC (fevereiro de 2016). "Selenocisteína: Por que estás?". Journal of Proteome Research . 15 (2): 677–8. doi : 10.1021 / acs.jproteome.5b01028 . PMID 26680273 .
links externos
| Scholia tem um perfil de tópico para selenocisteína . |
